Модернизация базы данных об анализах на ДНК микрочипах
Московский
государственный университет им. М.В. Ломоносова
Филиал МГУ в
г.Севастополе
Факультет
ВМиК
Курсовая
работа
МОДЕРНИЗАЦИЯ
БАЗЫ ДАННЫХ ОБ АНАЛИЗАХ НА ДНК МИКРОЧИПАХ
Выполнил: Терзи Иван,
Научный руководитель: Сальников А.Н.,
к.ф.-м.н.
г. Москва 2012 год.
Содержание
ВВЕДЕНИЕ
.
ИССЛЕДОВАНИЕ ПРЕДМЕТНОЙ ОБЛАСТИ
. ДНК-микрочипы.
Экспрессия гена. Способы хранения и обработки информации, полученной с помощью
ДНК-анализов
. СMSDrupal:
архитектура и особенности
. Сравнение
Drupal с другими CMS
. ПОСТРОЕНИЕ
МОДУЛЯ КОНТРОЛЯ И ДОСТУПА К БД ДНК-МИКРОЧИПОВ
. Описание
инструментов и средств разработки
. Структура
БД DNA Microarrays Access Control Interface
3. Структура модуля DNA Microarrays Access Control Interface
ЗАКЛЮЧЕНИЕ
СПИСОК
ИСПОЛЬЗОВАННОЙ ЛИТЕРАТУРЫ
ВВЕДЕНИЕ
Появление во 2-й половине 90-х годов ДНК-чипов стало новой
биотехнологической революцией, стоящей по значимости в одном ряду с
расшифровкой структуры ДНК в 50-х, исследованием фундаментальных
закономерностей молекулярной генетики. Учитывая огромную значимость этого
новейшего направления для развития современной фундаментальной и прикладной
медико-биологической науки представляется актуальным разработка программного
обеспечения для обработки больших массивов информационных данных. В текущий момент большую популярность
приобретает метод диагностики заболеваний на основе изучения процесса
экспрессии генов. На сегодняшний день в мире существует несколько публично
доступных баз данных значений экспрессии генов, которые хранятся как файлы
изображений полученных с соответствующих приборов. К таким базам можно отнести ArrayExpress, ExpressDB, GEO, SMD,
LAD,GENEVESTIGATOR, ArrayTrack, CIBEX.
Институт эпидемиологии и микробиологии имени Н.Ф. Гамалеи, выбранный
автором курсовой работы для сотрудничества, занимается исследованиями в области
раковых заболеваний с использованием анализов экспрессии генов ДНК-микрочипов.
На нынешний момент институт обладает примерно 10 000 проб с некоторыми данными
об этих пробах. Это обуславливает актуальность и необходимость разработки
удобного программного обеспечения, интерфейса администрирования и доступа к
базам данных ДНК-микрочипов пользователями интернет-ресурсов и сотрудниками
института.
Для решения этой задачи оптимальным на взгляд автора курсовой работы
является средство для разработки веб-интерфейсов, система управления (CMS) Drupal. Drupal является свободным программным обеспечением,
защищённым лицензией GPL, и развивается усилиями энтузиастов со всего мира, а
так же является одним из наиболее популярных программных продуктов. Архитектура
Drupal позволяет применять его для построения различных типов сайтов - от
блогов и новостных сайтов до информационных архивов или социальных сетей.
Имеющийся по умолчанию функциональность можно увеличивать подключением
дополнительных расширений - «модулей» в терминологии Drupal. Наиболее важные
функции, предоставляемые Drupal:
· единая категоризация всех видов содержимого (таксономия) - от
форумных сообщений до блогов и новостных статей
· широкий набор свойств при построении рубрикаторов: плоские
списки, иерархии, иерархии с общими предками, синонимы, родственные категории
· вложенность категорий любой глубины
· поиск по содержимому сайта, в том числе поиск по таксономии и
пользователям
· разграничение доступа пользователей к материалам (ролевая
модель)
· динамическое построение меню
· поддержка XML-форматов:
· вывод документов в RDF/RSS
· агрегация материалов с других сайтов
· BlogAPI для публикации материалов с помощью внешних
приложений
· авторизация через OpenID
· символьные осмысленные URL (иначе «человеко-понятные» - ЧПУ)
· переводы интерфейса сайта на разные языки, а также поддержка
ведения разноязычного контента
· возможность создания сайтов с пересекающимся содержимым
(например общей базой пользователей или общими настройками)
· раздельные конфигурации сайта для различных виртуальных
хостов (мультисайтинг), в том числе собственные наборы модулей и тем оформления
для каждого подсайта.
· уведомления о выходящих обновлениях модулей
На данный момент существует более 15 000опубликованных модулей,
написанных на Drupal. Модуль, разрабатываемый автором
данной работы,позволит обычным пользователям создавать, удалять, редактировать
наборы ДНК-проб, проверять соответствие набора проб чипу, удобно управлять
метаданными пациента, а так же выбирать пациентов по списку имеющихся проб.
Цель работы - Создание модуля Drupal для хранения информации с
результатами экспериментов по анализу экспрессии генов и её администрирования
(в рамках задачи разработки ПОдля БД анализов ДНК-микрочипов, предложенной
институтом эпидемиологии и микробиологии имени Н.Ф. Гамалеи).
Задачи работы:
1. Ознакомиться с теоретическими и эмпирическим аспектами, а так же
историей создания ДНК-микрочипов;
. Изучить структуры баз данных по пробам ДНК микрочипов (GEO,
Array Express а так же БД института Гамалеи Microarrays(cluster.gamaleya.org));
. Изучитьуже существующее программное обеспечение для работы с
данными полученными из ДНК микрочипов;
. Изучить язык PHP;
. Изучить синтаксис MySQL запросов;
. Изучить строение и структуру написания модуля на Drupal (Изучить систему управления сайтом
Drupal; Охарактеризовать её основные возможности; Изучить методы разработки
модулей на Drupal; Сравнить Drupalс другими CMS);
. Написать модуль для управления наборами пациентов и проб с
помощью Drupal-6, с помощью которого
можно:а)создавать, удалять, редактировать наборы проб, б)удобно управлять
метаданными пациента, в)выбирать пациентов по списку имеющихся проб.
1. ИССЛЕДОВАНИЕ ПРЕДМЕТНОЙ ОБЛАСТИ
1. ДНК-микрочипы. Экспрессия гена. Способы хранения и
обработки информации, полученной с помощью ДНК-анализов
В текущий момент большую популярность приобретает метод диагностики
заболеваний на основе изучения процесса экспрессии генов [2]. Массовая
диагностика на основе экспрессии генов в медицинских лабораториях становится
возможной благодаря появлению технологии создания так называемых
ДНК-микрочипов.
ДНК-микрочипы - технология, когда на подложку, из стекла или кремния в
определённые заранее позиции подложки наносится слой коротких от (20 до 60
нуклеотидов) одноцепочечных молекул ДНК. Последовательность нуклеотидов каждой
из молекул известна заранее. Эта последовательность чаще всего соответствует
одному из фрагментов исследуемого гена или одному из фрагментов известного
геномного повтора. Все подложки помещаются в специальный раствор, в котором
содержатся исследуемые окрашенные фрагменты молекул ДНК, которые связываются с
фрагментами на подложке по принципу комплиментарности.
В целом технология получения сведений с ДНК-микрочипа выглядит следующим
образом:
. Выделяются или выращиваются биологические образцы, которые
необходимо сравнить. Они могут соответствовать одним и тем же индивидуумам до и
после какого-либо лечения (случай парных сравнений), либо различным группам
индивидуумов, например, больным и здоровым, и т. д.
2. Из
образца выделяется очищенная нуклеиновая кислота, являющаяся объектом
исследования: это может быть РНК
<#"587506.files/image001.gif">
Рисунок 1: Пример изображения с ДНК-микрочипа
В текущий момент в мире существует несколько публично доступных баз
данных значений экспрессии генов, которые хранятся как файлы изображений
полученных с соответствующих приборов. К таким базам можно отнести:
1. ArrayExpress - база данных европейского биоинформатического института. В
этой базе хранятся данные об экспериментах с функциональной частью генома. В
частности об экспериментах с экспрессией генов. Данные в базе данных хранятся в
форматах MIAME и MINSEQE. Данные о экспрессии генов собраны в отдельную подбазу
Atlas [4], которая дополнительно курируется экспертами.
2. ExpressDB - база данных экспрессии генов бактерии кишечная палочка
(E.Coli). В этой базе данные заняться в собственном формате похожем на формат
MIAME. Используются ДНК-микрочипы компании Affymetrix первого поколения [5].
3. GEO (GeneExpressionOmnibus) - база данных является публичным
репозиторием для хранения данных полученных с микрочипов. Здесь хранятся как
данные о экспрессии генов, так и данные полученные с помощью современных
технологий секвенирования генома, которые основаны на технологии микрочипов.
Научному сообществу предоставляется возможность добавлять свои данные об
экспериментах, однако процесс добавления записей строго формализован и
тщательно курируется экспертами (подобно базе Atlas). Данные хранятся в MIAME
формате. База данных поддерживается Национальным Центром Информации о
Биотехнологии (NCBI) США. Информацию о базе данных GEO можно найти в статьях
[6], [7]. На момент 22 июля 2010 года в базе данных содержится 457879 записей.
В текущий момент это крупнейшая база данных результатов экспериментов с
ДНК-микрочипами и РНК-микрочипами.
4. SMD - Стенфордская база данных [8]. В отличии от описанных выше
баз данных содержит дополнительно закрытые данные коммерческих компаний.
Предоставляет набор программных средств для анализа и поиска информации в
микрочипах.
5. LAD (LonghornArrayDatabase) - клон Стенфордской базы данных, но
без данных коммерческих компаний. Эта база данных поддерживает формат записей
MIAME, построена на открытых технологиях (используется Linux и PostgreSQL ).
Информация о данной базе была опубликована в статье [9].
6. GENEVESTIGATOR - коммерческая база данных по экспрессии генов. Обладает
большим объёмом данных, однако за доступ к базе данных необходимо платить
деньги. Поддерживается двумя лабораториями в Швейцарии [10].
7. ArrayTrack - специализированная база данных содержит данные о экспрессии
генов для задач фармакологии и для анализа токсичности тех или иных продуктов
питания или других живых объектов. Данная база данных поддерживает MIAME
формат. Для того, чтобы ей воспользоваться необходимо устанавливать
дополнительное программное обеспечение. [11] Данная база поддерживается
(Национальным центром токсикологических исследований) министерством питания и
здравоохранения США.
8. CIBEX - база данных ДНК-микрочипов. Находится на территории Японии
строго поддерживает MIAME формат.
Очевидно, что на текущий момент наиболее популярным способом хранения
данных ДНК-микрочипов является способ хранения в формате MIAME. Спецификация
формата носит название MAGE-ТAB и была опубликована в журнале BMC
Bioinformatics в 2006 году [12]. Формат позволяет описывать эксперимент как
ориентированный ациклический граф, где вершина - одно действие эксперимента, действие
может быть выраженно как матрица с данными, а может соответствовать пустому
множеству сохранённых данных, а ребро - способ получения очередной матрицы с
данными. Пример такой концепции отображён на рисунке 2.
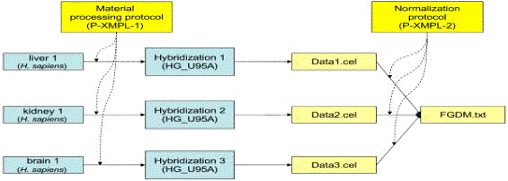
Рисунок 2. Шаги эксперимента (рисунок взят из статьи
На рисунке 2 в качестве источника выступают материалы взятые из разных
тканей человека, затем с ними производилась процедура гибридизации, то есть
образования химической связи между комплиментарными нуклеотидами, которые
«пришиты» к микрочипу, после чего мы видим файлы с результатами, которые будут
выражены матрицами значений, и затем среди этих файлов производится
нормализация, в результате чего получается конечный файл, который называется
«FGDM.txt». Весь процесс описывается в виде MAGE-TAB спецификации и состоит из
нескольких файлов.
Формат специфицирует несколько типов файлов:
• SDRF - файл, который задаёт привязку матриц, получающихся в
ходе эксперимента к их описанию. То есть по сути это описание шагов
приводящегося эксперимента.
• ADF - файл, с описаниями конкретных позиций микрочипа.
Например, какая в определённой позиции подложки «пришита» последовательность
ДНК и что она означает. В целом этот тип файлов обычно предоставляется
производителями ДНК-микрочипов.
• Raw - файл с данными. Это может быть текстовый или бинарный
файл. Файл может быть в формате предоставляемом производителем микрочипа.
Однако представлен также текстовый формат со значениями разделёнными символами
табуляции.
В файлах IDF, SDRF, ADF - значения разделяются символами табуляции,
текстовые поля должны быть заключены в двойные кавычки. В этих файлах допустимы
комментарии в виде строк начинающихся с символа '#'.
В IDF - файле должно присутствовать большое число полей. Опишем некоторые
из них:
• InvestigationTitle - название исследования.
• ExperimentalDesign - тип эксперимента по классификации MGED
• Experimental Design Term Accession
Number - идентификатортипаэксперимента
• ExperimentalFactorName - описание фактора который исследуется
в данном эксперименте. Используется в дальнейшем как идентификатор в SDRF
файле.
• ExperimentalFactorType - тип исследуемого фактора по
классификации MGED
• Experimental Factor Type Accession
Number - идентификатортипаисследуемогофактора.
• PersonLastName, PersonFirstName, PersonMidInitials - поля
описывающие имя человека имеющего отношение к представленным данным о
проводимом эксперименте.
• PersonEmail, PersonPhone, PersonFax, PersonAddress,
PersonAffiliation - адрес электронной почты для связи, телефон для связи, факс,
почтовый адрес, место работы соответственно.
• PersonRoles - описание роли человека в подготовке описываемых
форматом данных.
• QualityControlType - тип контроля за качеством получаемых в
эксперименте данных (по классификации MGED)
• ReplicateType - тип используемой репликации молекул по
классификации MGED. Обычно одна из разновидностей Полимеразной Цепной Реакции
(ПЦР).
• NormalizationType - тип нормализации результатов эксперимента
по классификации MGED.
• DateofExperiment - дата проведения эксперимента.
• PublicReleaseDate - это поле заполняется обычно
администраторами баз данных. Здесь указывается дата опубликования описываемых
форматом данных.
• PubMed ID, Publication DOI - идентификаторы статьи об
эксперименте в базах данных публикаций.
• Publication Author List, Publication
Title, Publication Status - информацияопубликации.
• ExperimentDescription - развёрнутое описание исследования
которое проводится в данном эксперименте.
• ProtocolName - название правила, по которому одни данные
будут преобразованы в другие. Это поле задаёт ребро в ациклическом графе
описывающем эксперимент. В дальнейшем значение будет использовано как
идентификатор в других типах файлов описываемых форматом.
• ProtocolType - тип правила преобразования данных (по
классификации MGED).
• ProtocolDescription - описание правила в свободной форме.
• ProtocolParameters - список параметров которые используются
правилом для выполнения действия. Параметры перечисляются через точку с
запятой. В дальнейшем каждый параметр будет использован как идентификатор.
• ProtocolHardware - описание оборудования, которое
используется для выполнения шага эксперимента при обработке данных с помощью
правила преобразования данных.
• ProtocolSoftware - описание программного обеспечения, которое
необходимо применить для выполнения шага эксперимента (например для
нормализации данных).
• SDRF File - список SDRF файлов связанных с данным IDF файлом.
• TermSourceName - список имён входных данных в дальнейшем
используются как идентификаторы.
• TermSourceFile - файл, где исходные данные записаны или это
может быть интернет ссылка (URL) на данные.
• Comment[<имя определяемое экспериментатором>] - это
поле используется вбазами данных для отображения прочей информации задаваемой
экспериментатором.
В IDF файле каждое поле пишется с новой строки. Сперва идёт имя поля,
затем табуляция и значение поля. В случае, если поле имеет несколько значений,
то значения должны быть разделены символом табуляции. ПоляInvestigation Title, Date
of Experiment, Public Release Date, Experiment Description должныиметьтолькооднозначение.
SDRF файл устроен как таблица значений: строка в таблице это
последовательность шагов эксперимента над одним источником данных. Столбцы в
таблице - действия, которые производятся над данными.. Причём считается, что
строки упорядочены по времени. То есть если источники данных обрабатываются в
определённом порядке, то строки в файле должны быть расположены именно в этом
порядке. Первая строка в файле - строка заголовок, остальные - строки значения.
Значения в столбце должны соответствовать заголовку. В заголовке поля
группируются в секции. Секция состоит из главного поля секции, после чего идёт
несколько подчинённых полей. Некоторые поля могут соответствовать параметрам,
которые были определены в IDF файле. В этом случае пишется имя поля с
параметром и имя параметра в квадратных скобках, например так: «Characteristics [occupancy]» Приведём далее описание некоторых
секций.
• SampleName - идентификатор материала с которым ведётся
эксперимент. Например «пробирка номер 234»
◦ Characteristics [<название характеристики>] - поле с
аргументом. Какая-то характеристика материала, характеристик может быть много
или они могут отсутствовать вовсе.
◦ Materialtype - указание типа материала с которым ведётся
эксперимент. Если это было РНК выделенное из всех клеток какого-либо организма,
то в значение нужно написать «whole_organism». В целом это поле должно быть
заполнено по классификации MGED.
◦ Description - вольное описание материала.
◦ Comment [<имя комментария>] - комментарий к материалу,
с дополнительным указанием имени комментария.
• HybridizationName - название способа, которым производится
присоединение фрагментов молекул ДНК или РНК к молекулам пришитым на подложку
микрочипа. В целом это поле может быть например именем технологии, например
«Affymetrix» или «Illumina».
◦ ArrayDesign REF - внешнее имя микрочипа, который применялся.
◦ Comment [<имя комментария>] - комментарий к микрочипу и
химическому процессу.
• Protocol REF - ссылка на описание протокола в IDF файле.
◦ ParameterValue[<имя параметра>] - список параметров был
указан в IDF файле. В SDRF файле хранятся значения.
◦ Performer - имя человека, который производил действие
определяемое протоколом.
◦ Date - дата проведения шага эксперимента.
◦ TermSource REF - ссылка на внешнее описание протокола.
◦ Comment [<имя комментария>] - типизированный
комментарий.
• ArrayDataFile, DerivedArrayDataFile, ImageFile - в значениях
этих полей должны быть написаны имена файлов. Файлы соответственно указанному
порядку это: необработанные данные с микрочипа, обработанные данные с
микрочипа, само изображение, которое было получено при фотографировании
микрочипа. Одной строчке в SDRF файле соответствует один файл.
◦ Comment [<имя комментария>] - типизированный
комментарий.
• ArrayDataMatrixFile, DerivedArrayDataMatrixFile - имя файла,
где могут храниться данные с более чем одного микрочипа. Также как и в
предыдущем случае один файл на одну строчку SDRF файла.
◦ Comment [<имя комментария>] - типизированный
комментарий.
Обратимся теперь к той части формата, которая описывает данные самих
микрочипов. Форматов хранения данных микрочипов существует достаточно много:
Generic, Affymetrix, GenePix, Agilent, ScanAlyze, ScanArray, QuantArray,
Arrayvision, Spotfinder, BlueFuse, UCSF Spot, NimbleScan, AppliedBiosystems,
CodeLink, Illumina, Imagene, CSIRO Spot. Рассмотрим только некоторые из них:
• Generic - это текстовый файл, где поля разделены символами
табуляции. В файле присутствуют поля MetaColumn, MetaRow, Column, Row - эти
поля задают координаты в получающемся после фотографирования подложки
изображении. Одинаковые значения полей MetaColumn и MetaRow чаще всего
соответствуют одному гену. Столбцы с координатами идут в начале, далее идут
столбцы со значениями уровня светимости микрочипа и другими характеристиками.
• GenePix - это формат схожий с Generic форматом, но здесь
присутствует добавочное поле Block. Блок содержит фрагменты подложки
принадлежащие одному гену.
• ImaGene - формат схожий с Generic, но в строке явно
присутствует GeneID.
Существуют
программы, которые облегчают импорт MAME файлов в базы данных. Например
tab2mage (#"587506.files/image003.gif">
3. Структура модуля DNA Microarrays Access Control Interface
При написании модуля автором использовалось огромное количество функций.
Приводим наиболее значимые из них:
Function dnamicro
arraysinter faceinstall()- функция создания базы данных,
создания ролей;
Function dnamicro
arraysinter faceuninstall()- функция удаления базы данных и
использованных таблиц;
Function dnamicro
arraysinter faceschema()-функция создания структуры БД;
Function dnamicro arraysinter facemenu()-функция создания меню; addition menu()- функция создания заголовков меню;
Function create new characteristic()- функция создания новых характеристик; dnamicro arraysinter face form()- функция создания форм заполнения и занесения в БД.
Так же использовались уже имеющиеся функции Drupal такие, как:
функция запросов к БД;
функция создания таблиц и др.
ЗАКЛЮЧЕНИЕ
В ходе данной курсовой работы были решены все поставленные задачи. Были
изучены теоретические и практические аспекты построения БД ДНК-микрочипов, а
так же рассмотрены существующие БД GEO, ArrayExpress и т.д. Была подробно изучена система
управления контентом Drupal, рассмотрены все её главные составляющие,
охарактеризованы основные возможности. Было проведено сравнение данной системы
с другими подобными CMS, выявлены их достоинства и недостатки. Также были
изучены методы создания модуля с помощью CMS Drupal, описана структура этого
модуля.
Таким образом, была достигнута цель настоящей курсовой работы -
разработан интерфейс контроля и доступа к БД ДНК-микрочипов с помощью CMS
Drupal. Для достижения этой цели использовались следующие средства разработки :
язык программирования PHP
система управления базами данных MySQL
Каждое из этих средств также было подробно рассмотрено.
В итоге была создан уникальный модуль для решения таких задач, как:
а) создание, удаление, редактирование наборов проб;
б) удобное управление метаданными пациента;
в) выборка пациентов по списку имеющихся проб.
Кроме того данная работа представляет возможность дальнейшей разработки
программного обеспечения в рамках БД института эпидемиологии и микробиологии
имени Н.Ф. Гамалеи. А именно:
1. Написать модуль для получения на кластер данных о наборах
пациентов группами
. Модернизировать имеющийся модуль для управления наборами
пациентов и проб, а именно:
1) Проверять соответствие набора проб чипу
) Строить множество чипов, содержащих данный набор проб
) Выбирать пациентов гибким поиском по метаданным, генерировать по
таким выборкам наборы пациентов
. Написать модуль для визуализации полученных данных в результате
работы и показывать их в веб-интерфейсе:. Гистограммы распределений значений
пробы в рамках набора пациентов. Ссылки для скачивания соответствующих
табличек
. Разработать интерфейс управления бинарными алгоритмами. Просмотр/редактирование
алгоритмов из веб-интерфейса. Применение алгоритма к пациенту/пациентам
из базы. Применение алгоритма к загруженным через форму
пациенту/пациентам
. Всем работающим скриптам проверять авторизованность пользователя
в друпале
СПИСОК ИСПОЛЬЗОВАННОЙ ЛИТЕРАТУРЫ
1. Т.
В. Наседкина «Использование биологических микрочипов в онкогематологии» //
Онкогематология, N1-2, стр. 25-37. 2006 г.
. И.К.
Гоголевская «Преимплантационная генетическая диагностика: современное состояние
и последние научные открытия» Материалы II международного конгресса по
преимплантационной генетике (18-24 сентября 1997г., Чикаго, США) // журнал
проблемы репродукции, N1, 1999 г. стр. 19-26.
. Айвазян
С.Р., Грановский И.Э., Филиппова В.В., Воронцова Н.И., Малов В.А., Белецкий
И.П. Современная лабораторная диагностика острых кишечных инфекций. //
Молекулярная медицина. - 2009.-№3.- с.3-8.
4. MishaKapushesky,
Ibrahim Emam, Ele Holloway, PavelKurnosov, AndreyZorin, James Malone, Gabriella
Rustici, Eleanor Williams, Helen Parkinson and AlvisBrazma «Gene Expression
Atlas at the European Bioinformatics Institute» // Nucleic Acids Research,
2010, Vol. 38, Database issue: pp. D690-D698.
5. Aach, J.,
Rindone, W., Church, G.M. (2000) Systematic management and analysis of yeast
gene expression data <http://arep.med.harvard.edu/pdf/Aach00.pdf> . GenomeResearch 10: 431-445.
<http://www.genome.org/cgi/content/abstract/10/4/431>
6. Tanya
Barrett and Ron Edgar Gene Expression Omnibus (GEO): Microarray data storage,
submission, retrieval, and analysis // Methods Enzymol. 2006; 411: 352-369.
7. Tanya
Barrett, Dennis B. Troup, Stephen E. Wilhite, Pierre Ledoux, Dmitry Rudnev,
Carlos Evangelista, Irene F. Kim, Alexandra Soboleva, Maxim Tomashevsky,
Kimberly A. Marshall, Katherine H. Phillippy, Patti M. Sherman, Rolf N.
Muertter, and Ron Edgar NCBI GEO: archive for high-throughput functional
genomic data //Nucleic Acids Res. 2009 January; 37(Database issue): pp. D885-D890.
8. Jeremy
Hubble, Janos Demeter, Heng Jin, Maria Mao, Michael Nitzberg, T. B. K. Reddy,
Farrell Wymore, Zachariah K. Zachariah, Gavin Sherlock and Catherine A. Ball
Implementation of GenePattern within the Stanford Microarray Database //Nucleic
Acids Research, 2009, Vol. 37, Database issue: pp. D898-D901.
9. Patrick
J Killion, Gavin Sherlock and Vishwanath R Iyer The Longhorn Array Database
(LAD): An Open-Source, MIAME compliant implementation of the Stanford
Microarray Database (SMD) // BMC Bioinformatics 2003, 4:32
. Tomas
Hruz, Oliver Laule, Gabor Szabo, et al., “Genevestigator V3: A Reference
Expression Database for the Meta-Analysis of Transcriptomes,” Advances in
Bioinformatics, vol. 2008, Article ID 420747, 5 pages, 2008.
. Davis,
A.P., Murphy, C.G., Saraceni-Richards, C.A., Rosentstein, M.C., Wiegers, T.C.,
Mattingly, C.J. Comparative Tosicogenomics Database: a knowledgebase and
discovery tool for chemical-gene-disease networks. NucleicAcidsRes2009,
37(Databaseissue): pp. D786-D792.
12. Tim
F Rayner, Philippe Rocca-Serra, Paul T Spellman, Helen C Causton, Anna Farne,
Ele Holloway, Rafael A Irizarry, Junmin Liu, Donald S Maier, Michael Miller,
Kjell Petersen, John Quackenbush, Gavin Sherlock, Christian J StoeckertJr,
Joseph White, Patricia L Whetzel, Farrell Wymore, Helen Parkinson, UgisSarkans,
Catherine A Ball and AlvisBrazma A simple spreadsheet-based, MIAME-supportive
format for microarray data: MAGE-TAB //BMC Bioinformatics 2006, 7:489
. Ash
A. Alizadeh, Michael B. Eisen, R. Eric Davis, Chi Ma, Izidore S. Lossos,
Andreas Rosenwald, Jennifer C. Boldrick, HajeerSabet, Truc Tran, Xin Yu, John
I. Powell, Liming Yang, Gerald E. Marti, Troy Moore, James Hudson, Jr, Lisheng
Lu, David B. Lewis, Robert Tibshirani, Gavin Sherlock, Wing C. Chan, Timothy C.
Greiner, Dennis D. Weisenburger, James O. Armitage, Roger Warnke, Ronald Levy,
Wyndham Wilson, Michael R. Grever, John C. Byrd, David Botstein, Patrick O.
Brown and Louis M. StaudtDistinct types of diffuse large B-cell lymphoma
identified by gene expression profiling // Nature 403, 2000, pp. 503-511.